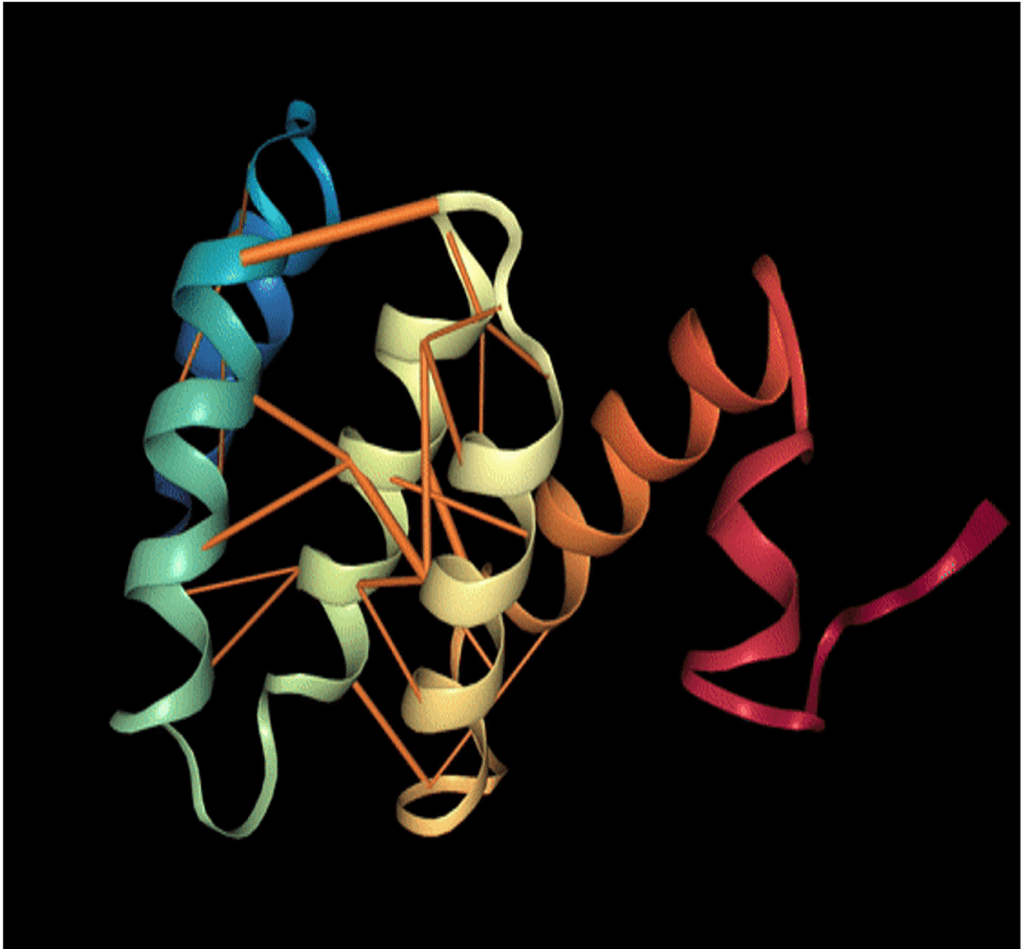Cela fait maintenant plus d’un an que le monde entier doit affronter un virus de type Sars-Cov-2. Sa vitesse de propagation ainsi que la diversité des zones géographiques touchées l’ont rapidement classé en tant que pandémie. Pour faire face à cette crise sanitaire, les recherches se sont multipliées dans tous les secteurs scientifiques avec un double enjeu : trouver un traitement mais aussi limiter la propagation de l’épidémie. C’est là que le Natural Language Processing est entré en jeu dans la lutte contre le COVID-19. Quel rôle le NLP a-t-il pu jouer dans l’élaboration d’un traitement ? Comment a-t-il permis de limiter les conséquences néfastes de la pandémie ? Comment a-t-il été utilisé pour estimer l’impact de la crise ? On vous explique tout dans cet article.
Par Guillaume SCHALLER, Data Scientist chez LittleBigCode
Les fonds alloués à la recherche associés à une collaboration publique et privée ont ainsi permis de combattre de façon inédite une pandémie de cette échelle. Pour illustrer cet effort mondial, le 1er février 2021, on recensait plus de 105 000 publications scientifiques selon le site COVID-19 Primer. En parallèle, l’intelligence artificielle a obtenu des résultats très convaincants pour son utilisation dans notre quotidien, facilitée en ce sens par la quantité de données accessibles et la capacité de calcul des ordinateurs.
Le Natural Language Processing (NLP), branche majeure de l’intelligence artificielle, a grandement contribué à cette révolution par son large spectre d’application (traduction automatique, résumé, extraction de sentiments). Des modèles NLP qui se sont eux aussi grandement améliorés durant ces dernières années. À titre exemple, ces derniers sont souvent évalués sur GLUE (General Language Understanding Evaluation) qui constitue une collection de problèmes linguistiques variés (inférence, paraphrase, similarité entre des textes…) : DeBERTa de Microsoft y a obtenu un score général de 90,8 alors que la baseline n’était que de 70 il y a deux ans. Voilà pourquoi l’intelligence artificielle a permis de répondre à cette crise sanitaire d’une façon sans précédent.
1/ Le NLP pour décrypter le langage de la vie
Le premier cas d’usage du NLP s’est porté sur la recherche d’un traitement contre la COVID-19. Comment ? En utilisant les mêmes modèles qui servent à analyser notre langage, les scientifiques ont étudié les séquences de protéines afin de pouvoir déterminer en quelques semaines le squelette génétique du virus.
Pour simplifier, une protéine est une séquence d’une longueur variable composée d’environ 20 acides aminés. De ce point de vue, les protéines peuvent être considérées comme n’importe quel langage avec son alphabet (les acides aminés) pour former des phrases (la séquence primaire de la protéine). Or, s’il est presque impossible pour nous humains d’apprendre à parler la protéine, les modèles de NLP, en analysant des milliards de séquences de protéines associées à leur propriété, peuvent en comprendre la grammaire.
Des bases publiques comme Uniprot ont permis ces études. Cependant, ces données sont extrêmement coûteuses à labeliser car elles nécessitent des connaissances particulièrement poussées. Les modèles employés pour pouvoir utiliser cette richesse de données sont donc des modèles de self-supervised learning.
2/ Les modèles basés sur les Transformers
De quoi s’agit-il ? L’idée des Transformers est la même que celle ayant entraîné la naissance des modèles RNN (Recurrent Neural Network). Le traitement de l’entrée doit dépendre de l’entrée précédente . La principale limite de ces modèles RNN est qu’ils ont une fenêtre de contexte très limitée, ce qui les empêche de comprendre les relations qu’il peut y avoir entre des entrées très éloignées dans la séquence. Sachant que les protéines ont des séquences moyennes qui dépassent les 300 tokens, il est normal que les RNN soient peu utilisés pour étudier les séquences de protéines.
A) Le modèle Transformer
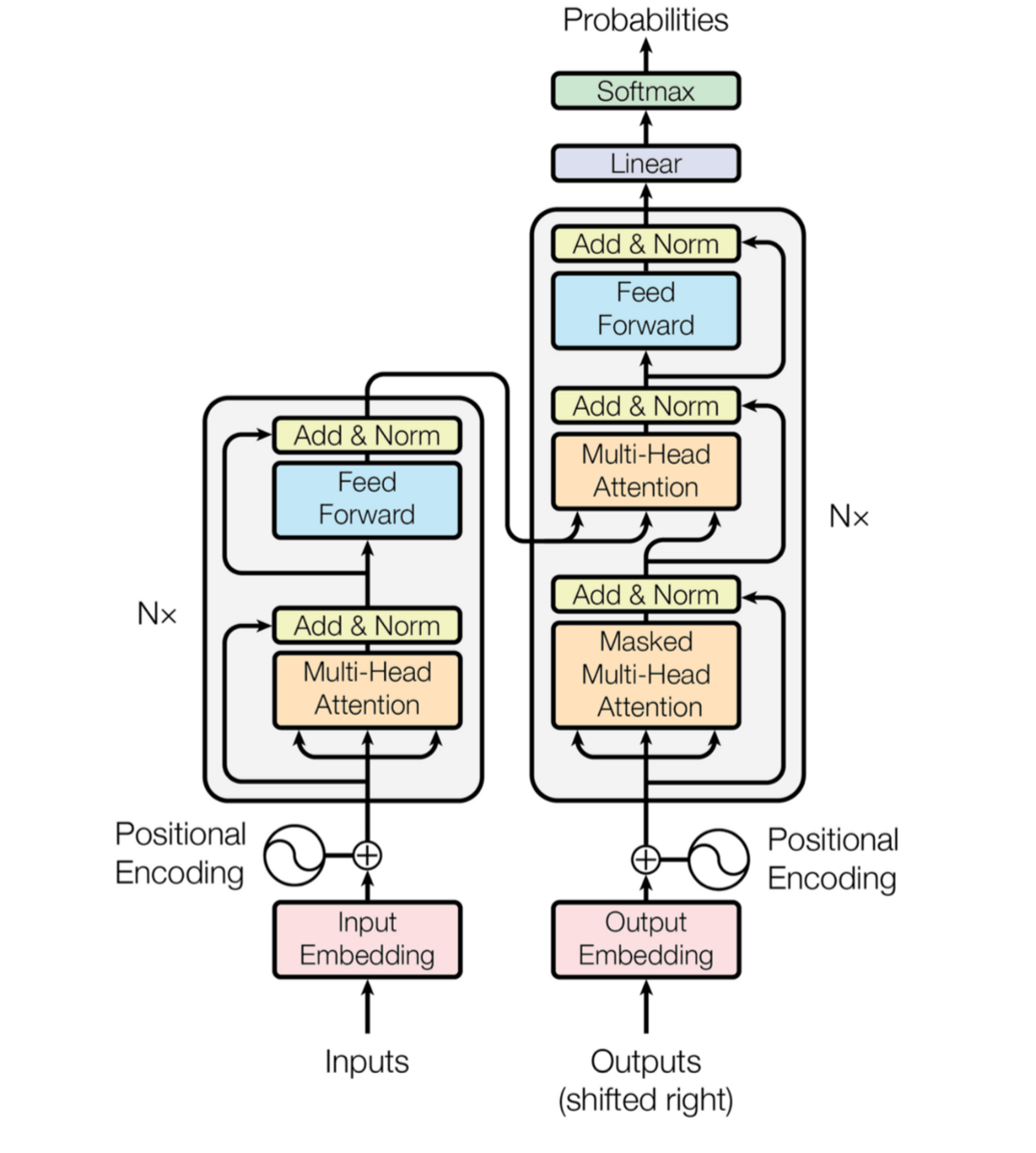
Les modèles Transformers de l’article scientifique Attention is all you need étudient les entrées en fonction d’un contexte mais sans avoir la moindre couche de neurones récurrents. Ces modèles utilisent des couches d’attention pour comprendre les dépendances entre les entrées à différentes positions.
FIGURE 1 : schéma d’un transformer (source : Attention is all you need)
Comme vous pouvez l’observer sur le schéma, l’encoder et le decoder empilent des couches de multi-head attention et de feed-forward associées à l’encoding de la position des mots. Objectif : aider le modèle à garder une compréhension de l’ordre de la séquence. De plus, on peut noter l’utilisation des couches de normalisation afin d’éviter le ‘‘vanishing gradient’’ dû au nombre important de produits de matrices dans la back-propagation.
B) Le mécanisme d’attention dans les séquences de protéine
Les protéines, comme expliqué précédemment, sont des séquences d’acides aminés qui forment une séquence primaire. Les interactions entre les différents blocs d’une séquence créent des formes (hélices…) qui sont la structure secondaire d’une protéine. Enfin, ces formes interagissent pour composer la structure tertiaire de la protéine.
Afin d’obtenir plus d’interprétation dans les résultats obtenus par les modèles de NLP sur les protéines, Rajesh Bhasin et Abhishek Kumar, dans leur article ProVis : An Anaglyph based Visualization Tool for Protein Molecules, ont couplé le poids d’attention du modèle des différents acides aminés avec la structure tertiaire de la protéine. En effet, chaque couche d’attention produit une distribution de probabilité w où représente le poids d’attention entre le token et . Plus ce poids est important, plus ces tokens sont dépendants.
Dans cet article scientifique, on réalise que des acides aminés très éloignés dans la séquence primaire en entrée dans le modèle peuvent avoir une grande dépendance. Celle-ci est du fait expliquée par leur proximité en 3D après la formation de la structure tertiaire de la protéine. Ainsi, sans avoir le moindre indice sur la structure physique de la protéine, les modèles de NLP ont pu capter l’interaction forte entre deux acides aminés proches dans l’espace.
FIGURE 2 : Protéine en 3D avec les mécanismes d’attention oranges provenant d’un modèle entrainé sur leurs séquences primaires 1D. Les cylindres oranges sont les poids d’attention DD dont le diamètre est proportionnel à la valeur.
C) L’évaluation des modèles
Mais la visualisation des mécanismes d’attention n’est pas tout. Il faut aussi savoir évaluer rapidement la modélisation du langage des protéines avec des métriques qui ont du sens, à la fois au niveau NLP et aussi au niveau biologique.
Dans un premier temps, tout comme pour un langage ordinaire, les modèles peuvent être évalués sur leur perplexité. Ce score, qui est une fonction de la cross-entropie d’une séquence, mesure la faculté de généralisation de la grammaire d’un langage. En effet, si la perplexité d’un mot est élevée, cela signifie que le modèle trouve qu’il n’est pas du tout adapté dans la séquence. Alors qu’un modèle avec une faible perplexité aura appris les nuances d’un langage. Ainsi, le fait de voir un nouveau mot mais de sens similaire ne le surprendra pas.
C’est pourquoi, on cherche à avoir des modèles de représentation du langage avec une perplexité faible. Une autre métrique envisageable repose sur la précision d’un modèle à prédire un token qui aurait été masqué.
En ce qui concerne les métriques intéressantes pour les biologistes, on peut évaluer un modèle de représentation du langage sur un corpus de tâches bio-informatiques. Présenté dans l’article scientifique Evaluating Protein Transfer Learning with TAPE | bioRxiv, ce corpus est une analogie avec la méthode d’évaluation de modèles de NLP sur un langage classique GLUE.
TAPE fournit 5 tâches avec leurs datasets associés pour benchmarker un modèle par rapport à l’état de l’art. Ces 5 tâches (Secondary Structure Prediction, Contact Prediction, Remote Homology Detection, Fluorescence Landscape Prediction et Stability Landscape Prediction) répondent toutes à des problématiques de modélisation de protéines.
Par exemple, la tâche « Secondary Structure Prediction » vise à évaluer la capacité d’un modèle n’ayant étudié que des séquences primaires à comprendre des propriétés liées à la forme secondaire des protéines. Cette tâche est donc un problème de séquence à séquence dans laquelle le modèle doit prédire pour chaque acide aminé sa forme dans la seconde structure (trois labels possibles : hélices, fils ou autres).
La tâche « Stability Landscape Prediction » est aussi un problème de régression de séquence à séquence qui vise à tester si les modèles apprennent à mesurer la stabilité des protéines. C’est extrêmement utile pour la recherche de création de protéines fonctionnelles car on peut facilement garder les mutations ayant le plus de chance de rester stables.
D) La génération de protéines pour la recherche de traitements
L’intérêt de la génération de protéines est avant tout représenté par le gain de temps qu’elle génère. Précédemment, la création de nouvelles séquences de protéines était établie sur la réalisation de mutations sur des parties de la séquence d’une protéine. L’idée était alors d’obtenir une propriété recherchée avec des techniques biologiques telle que l’évolution directe.
Néanmoins, pour une séquence de longueur L composée de m acides aminés, on a O( ) possibilités de mutations. Chaque mutation est longue et cher à réaliser.
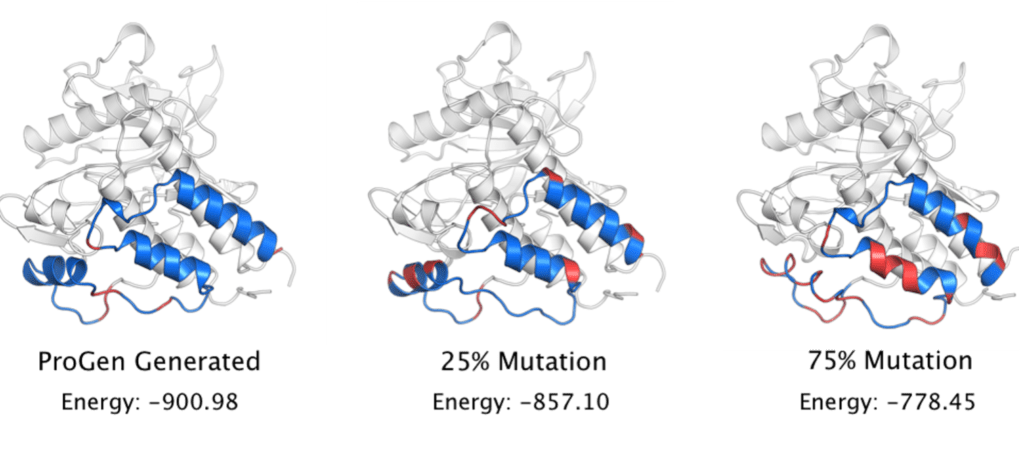
Des articles comme ProGen: Using AI to Generate Proteins (einstein.ai) proposent donc d’imiter ce fonctionnement. Ils modélisent le langage des protéines puis sélectionnent des séquences de protéines connues pour avoir certaines propriétés désirées. Ensuite, ils cachent des parties de la séquence et génèrent avec leur modèle les séquences manquantes. Les candidats générés ayant les meilleures propriétés biophysiques peuvent alors être recréés en laboratoire. Ils ont ainsi démontré que leurs séquences générées étaient fonctionnelles car elles conservaient les mêmes structures secondaires que les séquences initiales et minimisaient l’énergie interne. Afin de confronter leurs résultats avec des mutations aléatoires, ils ont alors créé des baselines en modifiant aléatoirement 25 et 75 % de la séquence avec des aminoacides au hasard.
FIGURE 3 : Séquence de protéines générées. La conservation de la structure secondaire est soulignée par l’énergie interne (faible en BLEU, haute en ROUGE). Source : Blog Einstein
Dans cette première partie de l’article, nous avons vu comment le NLP avait pu être utilisé pour identifier le mécanisme du virus et aider à élaborer un traitement. Dans la seconde partie, voyons comment le natural Language Processing a également permis de limiter les conséquences de la pandémie.